Um die Sequenz eines Gens herauszufinden, also die Abfolge seiner Buchstaben, muss das Gen zuerst kopiert werden. Wie das funktioniert, kannst du im Artikel Gene kopieren: Die Polymerase-Kettenreaktion nachlesen. Für die Sequenzierung wurden in den letzten 20 Jahren verschiedene moderne Techniken entwickelt. Viele wichtige Gen-Sequenzen fand man aber bereits mit der klassischen Methode, die hier als Beispiel vorgestellt wird (eine Weiterentwicklung der Sanger-Sequenzierung, benannt nach Frederick Sanger).
Für die Sequenzierung braucht es:
- Viele Kopien des Gens, das buchstabiert werden soll.
- Primer: kurze, einsträngige DNA-Stücke, die mit dem Anfang des Gens identisch sind.
- DNA-Polymerase (das „Kopiergerät“).
- Viele Bausteine A, C, G und T.
- Eine kleine Menge „Stop-Bausteine“, die wir hier ddA, ddC, ddG und ddT nennen. Die vier verschiedenen Stop-Bausteine sind mit unterschiedlichen fluoreszierenden Farbstoffen markiert.
Dieser Ansatz wird in einem kleinen Proberöhrchen zusammengemischt. Genau wie bei der Polymerase-Kettenreaktion stellt man das Röhrchen danach in ein Heizgerät mit einem vorprogrammierten Temperatur-Zyklus:
- 94°C: Die beiden DNA-Stränge des Gens trennen sich.
- 60°C: Die Primer heften sich an den Anfang der DNA-Einzelstränge
- 72°C: Die DNA-Polymerase beginnt zu arbeiten und ergänzt Baustein für Baustein den zweiten DNA-Strang.
Beim Kopiervorgang verwendet die DNA-Polymerase hauptsächlich die normalen Bausteine A, C, G und T, aber manchmal erwischt sie stattdessen einen der wenigen Stop-Bausteine. Dann bricht der Kopiervorgang an dieser Stelle ab, und das Gen wird nicht vollständig kopiert.
Unterschiedlich lange DNA-Kopien entstehen
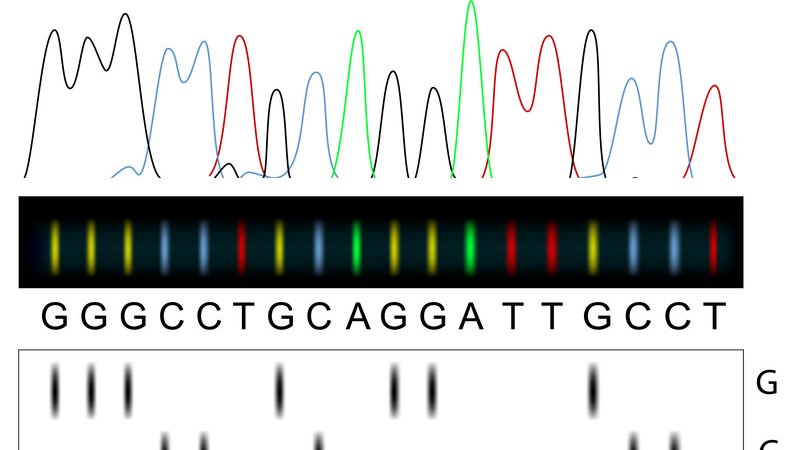
So entstehen unterschiedlich lange Bruchstücke des Gens, die alle gleich anfangen (mit dem Primer), aber an unterschiedlichen Stellen aufhören (dort, wo ein Stop-Baustein eingebaut wurde). Der Kopiervorgang wird sehr viele Male wiederholt – so lange, bis jeder Baustein des Gens einmal zufällig durch einen Stop-Baustein ersetzt wurde. Zu diesem Zeitpunkt hat man sehr, sehr viele DNA-Stücke in der Probe. Pro Buchstabe des Gens ist darunter mindestens ein DNA-Stück, dass vom Primer bis genau zu diesem Buchstaben reicht.
Der Grösse nach sortieren
Ähnlich wie bei einer Gel-Elektrophorese werden die DNA-Stücke nun der Länge nach sortiert. Es wird dazu aber ein sehr feines Röhrchen verwendet, das sogar DNA-Stücke trennen kann, die sich in der Länge nur um einen einzigen Baustein unterscheiden. Die kleinsten DNA-Stücke wandern am schnellsten durch das Röhrchen. Am Ausgang des Röhrchens befindet sich ein Detektor, der die Fluoreszenz-Farben der Stop-Bausteine ddA, ddC, ddG und ddT unterscheiden kann.
Nun lässt man die Probe mit den Gen-Kopien durch das Röhrchen laufen. Als erstes erscheinen die ganz kurzen Stückchen am Detektor, bei denen direkt nach dem Primer ein Stop-Baustein eingebaut wurde. Dann folgen nach und nach die nächsten, die immer genau einen Baustein länger sind als die vorherigen. Und da jeder Stop-Baustein eine unterschiedliche Farbe hat, kann der Detektor bei jeder Gruppe von DNA-Stückchen „lesen“, mit welchem Baustein oder Buchstaben sie aufhören. So buchstabiert man der Reihe nach das ganze Gen durch.